Grazie a strumenti come processing è possibile sperimentare ed esplorare in maniera abbastanza intuitiva nuovi sistemi che relazionino oggetti e comportamenti tra loro. Basta avere qualche nozione di programmazione ed integrandola con i tanti riferimenti/tutorial e documentazione online si riescono a fare autonomamente diversi passi avanti.
Il mio studio parte dalla comprensione di quelli che sono gli agent-system e i sistemi di flocking applicati nel campo specifico del micelio. Ognuna delle ife che va a costituire il micelio è difatto interpretabile come un singolo agente capace di diramarsi (branching ) e di esplorare lo spazio in funzione della quantità di cibo, degli agenti vicini, di un campo vettoriale.
Ringrazio co-de-it per aver condiviso il loro lavoro; punto di partenza fondamentale:
CTRL.SHIFT – the Control Shift | GH+P5 Co-de-iT workshop Vienna – 02>07 April 2013
Di seguito i vari step con i relativi progressi.
0.1
Il sistema di flocking solitamente non è pensato per potersi diramare, bensì è un insieme fisso di agenti che si muovono nello spazio. Dunque la prima modifica da fare era permettere ad ognuna delle ife di potersi diramare. Così a partire da un punto iniziale (spora) si è introdotta una variabile “probabilità di braching” che ha permesso di poter variare (inizialmente in maniera manuale) la possibilità dell’ifa di diramarsi. Quando viene attivata la funzione toBranch(); nasce una nuova ifa a partire dall’ultima posizione di quella designata a diramarsi.
0.2
Lo spazio finito in cui far partire la simulazione è costituito da un insieme discreto di punti ognuno dei quali possiede diversi attributi oltre a quella classica del punto (posizione). Questi attributi sono volti all’interpretazione del substrato ovvero la porzione di spazio reale in cui il micelio ha la possibilità di crescere. Si è aggiunto quindi a questi punti l’informazione sulla quantità di cibo presente e dell’influenza (tramite vettore) di un campo che ne indirizzi la crescita.
Questi valori sono caricati da file esterni e mi concentrerò più avanti sul definire il come vengono costruiti i vari substrati.
La probabilità di branching è stata dunque messa in relazione al substrato e alla quantità di cibo: più cibo c’è più la rete di ife si infittisce.
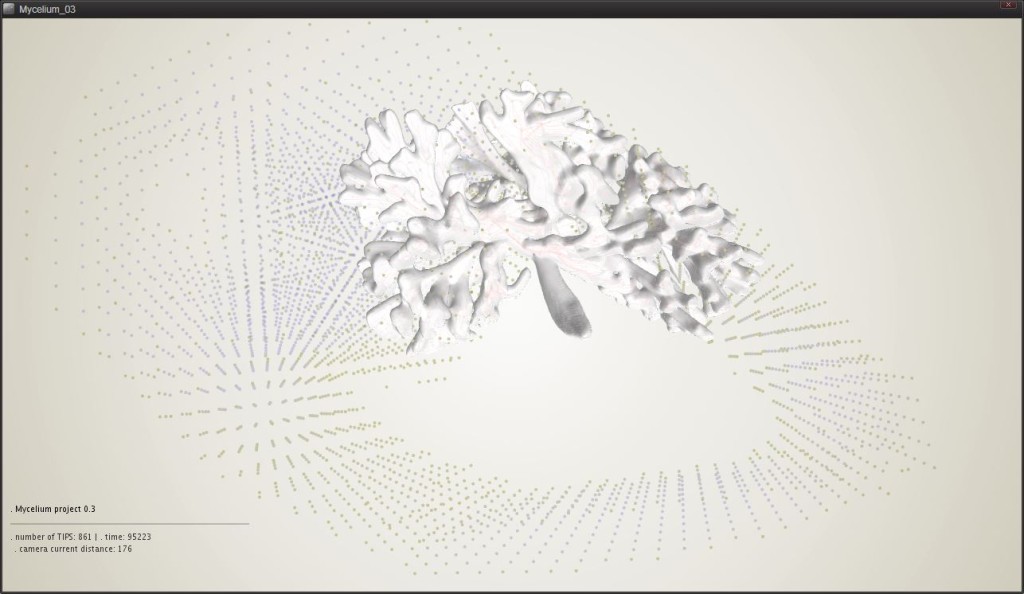
0.3
Dalla serie di ife viene poi implementata la possibilità di creare una mesh i cui punti sono funzione delle tracce lasciate dagli agenti in movimento. Immediato è il riscontro formale.
0.4
La possibilità di branching diventa fondamentale se contrastata dalla “morte” degli agenti. Ogni ifa dunque presenta uno stato alive e uno dead: muore quando la quantità di cibo all’interno di un determinato raggio è minore di una soglia fissata.
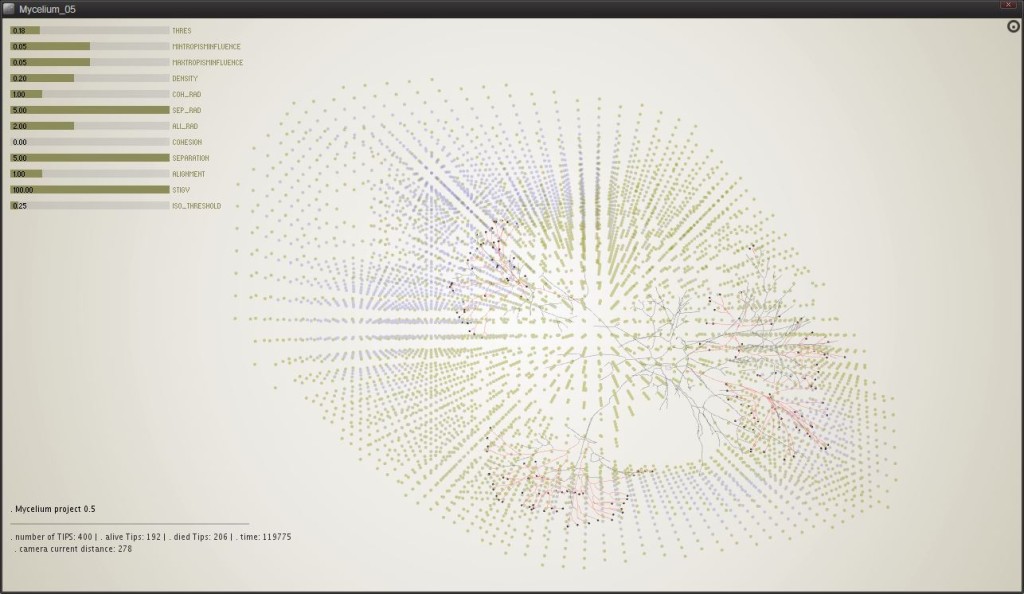
0.5
Non solo il micelio è influenzato dal substrato ma anche esso va a modificarsi dopo il passaggio delle ife in quello che può essere considerato un comportamento stigmergico: i nutrienti diminuiscono, i punti cambiano colore sino a scomparire.
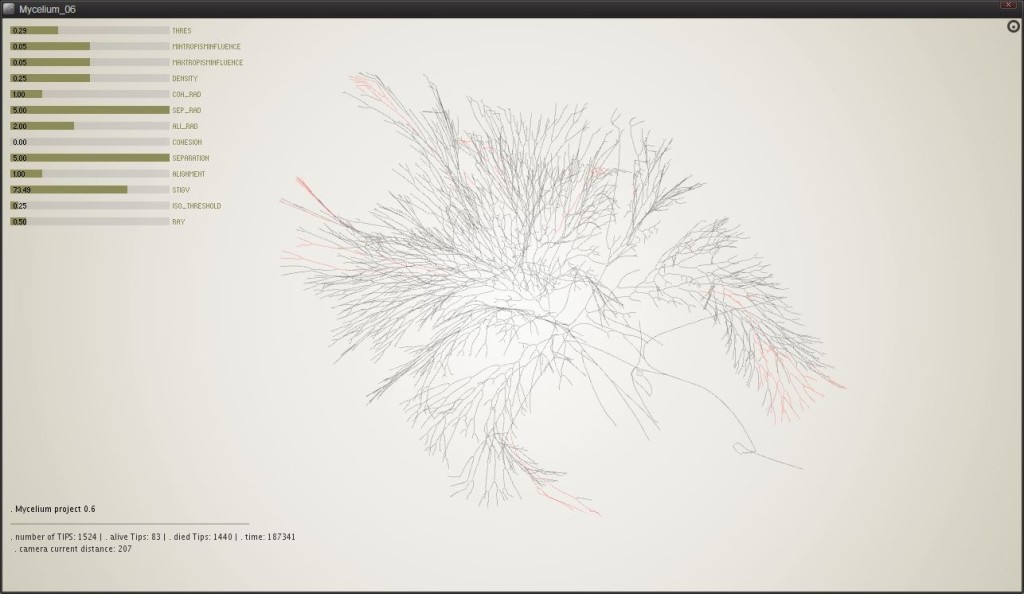
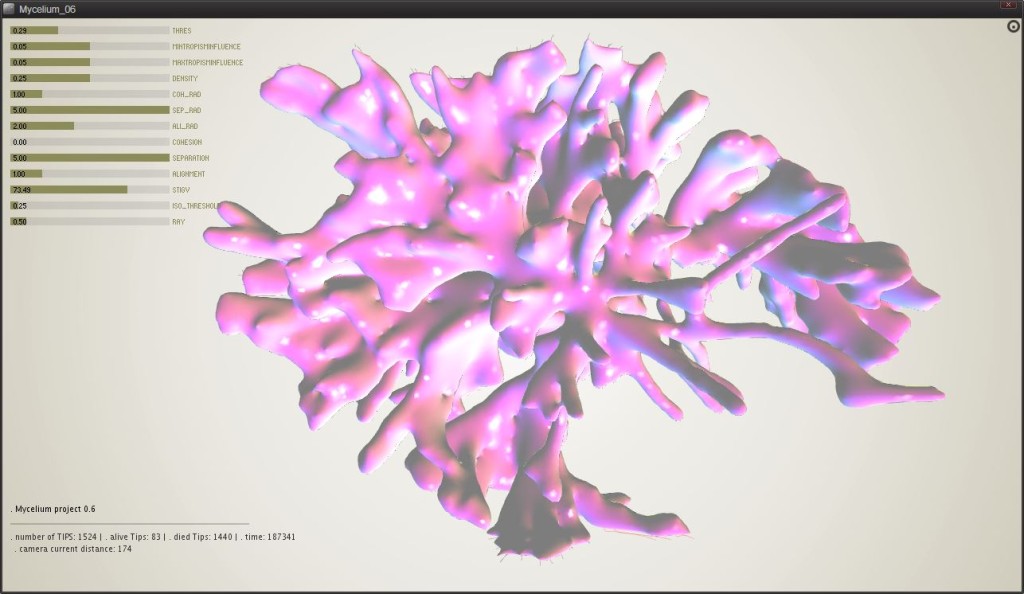
0.6
Con l’aggiunta di alcuni parametri, come ad esempio la possibilità di cambiare il raggio che definisce lo spazio in cui ogni singolo agente legge l’ambiente, iniziano ad emergere alcune proprietà adattive del sistema nonchè i primi risultati interessanti.
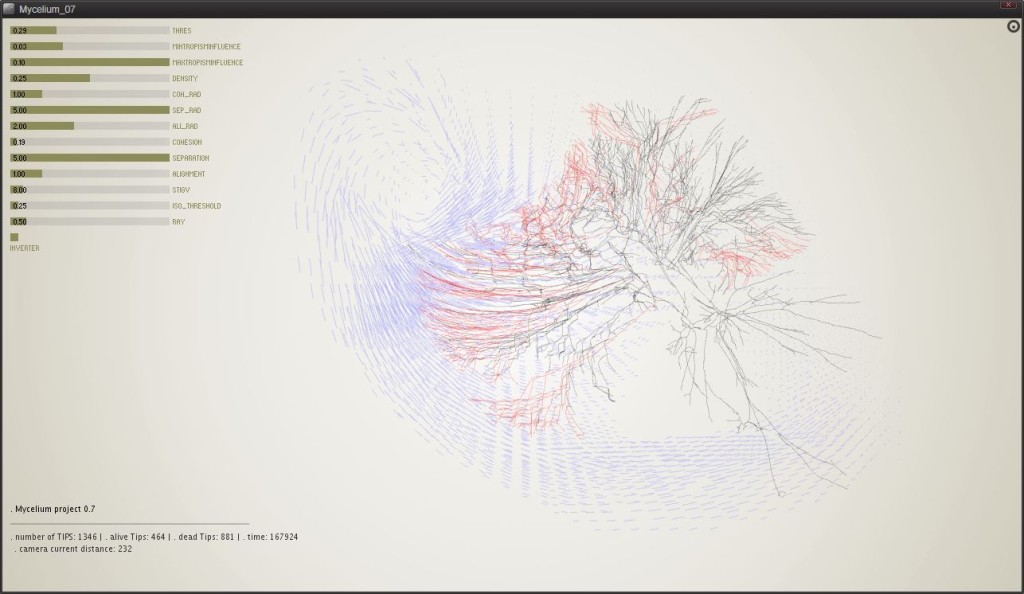
0.7
Aumentando la complessità si è qui introdotta l’influenza di un campo vettoriale sul tropismo delle varie ife. Regolabile in un dominio [minTropismInfluence : maxTropismInfluence]
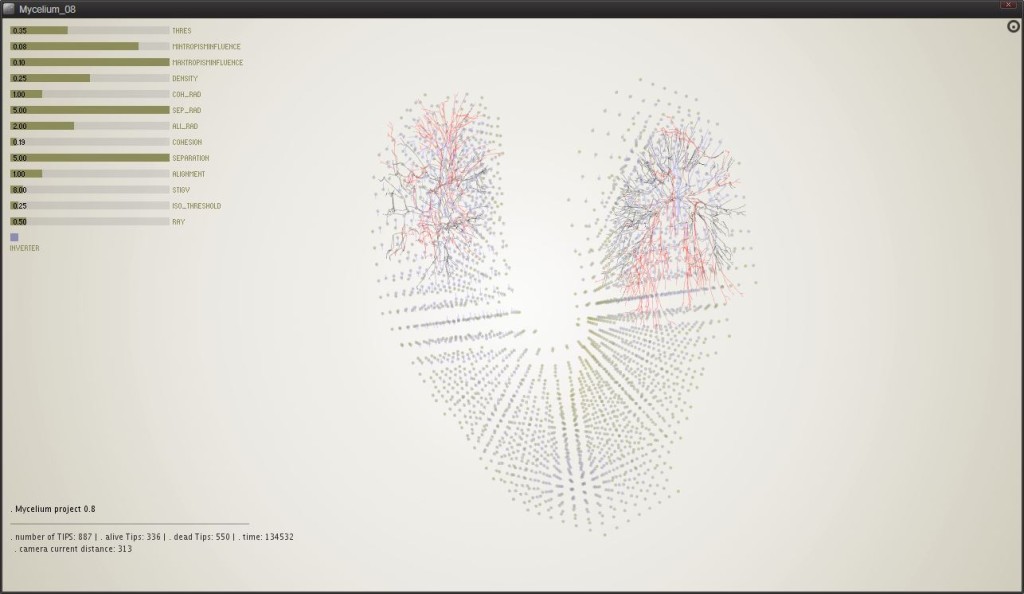
0.8
Le future implementazioni individuano lo studio di substrati differenti e la possibilità di avere più spore contemporaneamente (quindi più punti iniziali della simulazione).




Good afternoon gianluca.
Myself and some colleagues at London Metropolitan University have started a small mycological growth operation in our school. Our research is mostly focused around the latter growth phases and mould creation but we would like to start investigating the rhizomatic growth. If at all possible would you be available to have chat or exchange emails so that we can further discuss your work.
Thanks,
C
Hello Mr. Scully,
if you want to contact me, please feel free to write me. My email is at the top of this page, under my avatar.
Regards,
-g